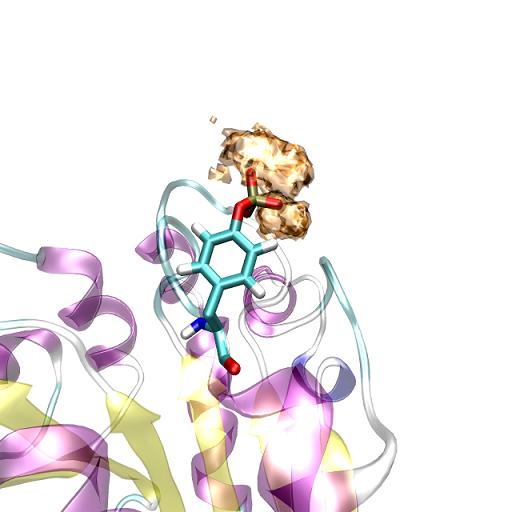
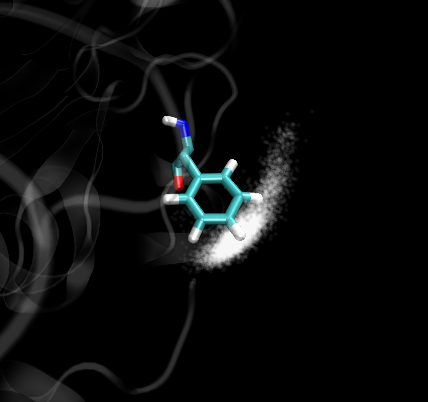
VMD显示轨迹中粒子的空间密度分布的两种方法
Two methods for VMD to display the spatial density distribution of particles in trajectories
文/Sobereva
第一种方法是用颜色深浅显示某原子在不同位置出现的概率,这种方法适合描述少数几个粒子的密度分布,效果比较柔和。
渲染模式开GLSL,在Materials对话框里把Transparent材质复制到一个新的材质叫a
把a的Ambient设最大,Opacity设0.03,然后在文本控制台source一下dis.tcl脚本。
运行draw material a
然后比如想看index 444原子在前4444帧内的空间分布状况,输入dis 444 4444。如果效果不好,再调a材质的Opacity。
原理简单来说,就是开了GLSL渲染模式之后所绘的东西可以有透明色,每次出现用一个透明的白色球来表示,这样的东西叠加次数越多,某处越不透明,显得颜色越深(图中的例子就是越白),说明出现概率越大。我的例子是苯丙氨酸苯环末端的H在各个位置的出现几率,说明不易偏离其稳定位置,越靠边概率越小。
球半径0.13是我觉得图像效果比较好的情况,可以自己调。如果不满意,就运行draw delete all删除所绘的一切内容,调完参数再次重复上面步骤运行。
dis.tcl脚本内容:
proc dis {atom fps} { draw color white for {set i 0} {$i<$fps} {incr i 1} { set nowselect [atomselect top "index $atom" frame $i] set x [$nowselect get {x}] set y [$nowselect get {y}] set z [$nowselect get {z}] draw sphere "$x $y $z" radius 0.13 } }
第二种方法是用等值面显示,这种方法适合描述大量粒子的空间分布。用g_spatial可以计算某个group在空间中每个点的出现几率。
首先用trjconv -f a-sol.trr -s a-sol.gro -fit rot+trans按照某个东西align一下,比如研究溶剂在蛋白附近分布,fit组就选蛋白,也就是以蛋白不发生整体运动为依据改变整个轨迹中的原子坐标,然后g_spatial -s a-sol.tpr -f a-sol.trr -n index.ndx可以计算某个group在空间中每个点的出现几率,存为grid.cube。如果说内存分配不够或者段错误,用-nab,设100一般够了。设bin控制格点宽度,默认是0.05,0.03的时候文件已经很大了。
g_spatial第一次选择的group决定了空间格点各个值的大小,也即决定了grid.cube的数据部分,得到的等值面图是什么样完全取决于第一次选择的group。第二次选择group实际上是选择输出哪些原子到grid.cube头部的原子叙述部分,这个完全不影响结果,随便设,一般设成溶质。比如第一组设的是溶剂,第二组设的是溶质,读进VMD,用CPK等模式看到的是溶质的结构,用isosurface模式看到的是溶剂出现密度的等值面。两个模式叠加显示就是溶剂与溶质的关系。